CRISPR/Cas9是一种快速有效的基因编辑工具,但是它在识别DNA时需要目标序列下游有一个特定的序列,称为PAM。PAM越短,在基因中分布就越多,Cas9能编辑的位点就越多。因此,开发识别短PAM的Cas9是研究热点。SaCas9是小型Cas9,能够被单个AAV病毒载体包装递送用于体内基因治疗。SaCas9识别的PAM是NNGRRT,受到4个碱基的限制,在基因组中分布较少。有研究人员将SaCas9识别的PAM改造成了NNNRRT,但是依然受到3个碱基的限制。王永明课题组先前鉴定了16个SaCas9同系物的PAM序列,其中SauriCas9和SlugCas9识别NNGG PAM,只受到2个碱基的限制。
2022年11月,王永明课题组又有新突破,联合东南大学柴人杰团队和苏州大学胡士军团队在PLOS biology上发表了题为“Identification of SaCas9 orthologs containing a conserved serine residue that determines simple NNGG PAM recognition”的研究论文。该研究分析了Cas9序列和PAM的对应关系,发现识别NNGG PAM的Cas9存在3个保守的氨基酸。如果将其中两个氨基酸替换到SaCas9上面,就能使SaCas9识别NNGG PAM(图1)。
|
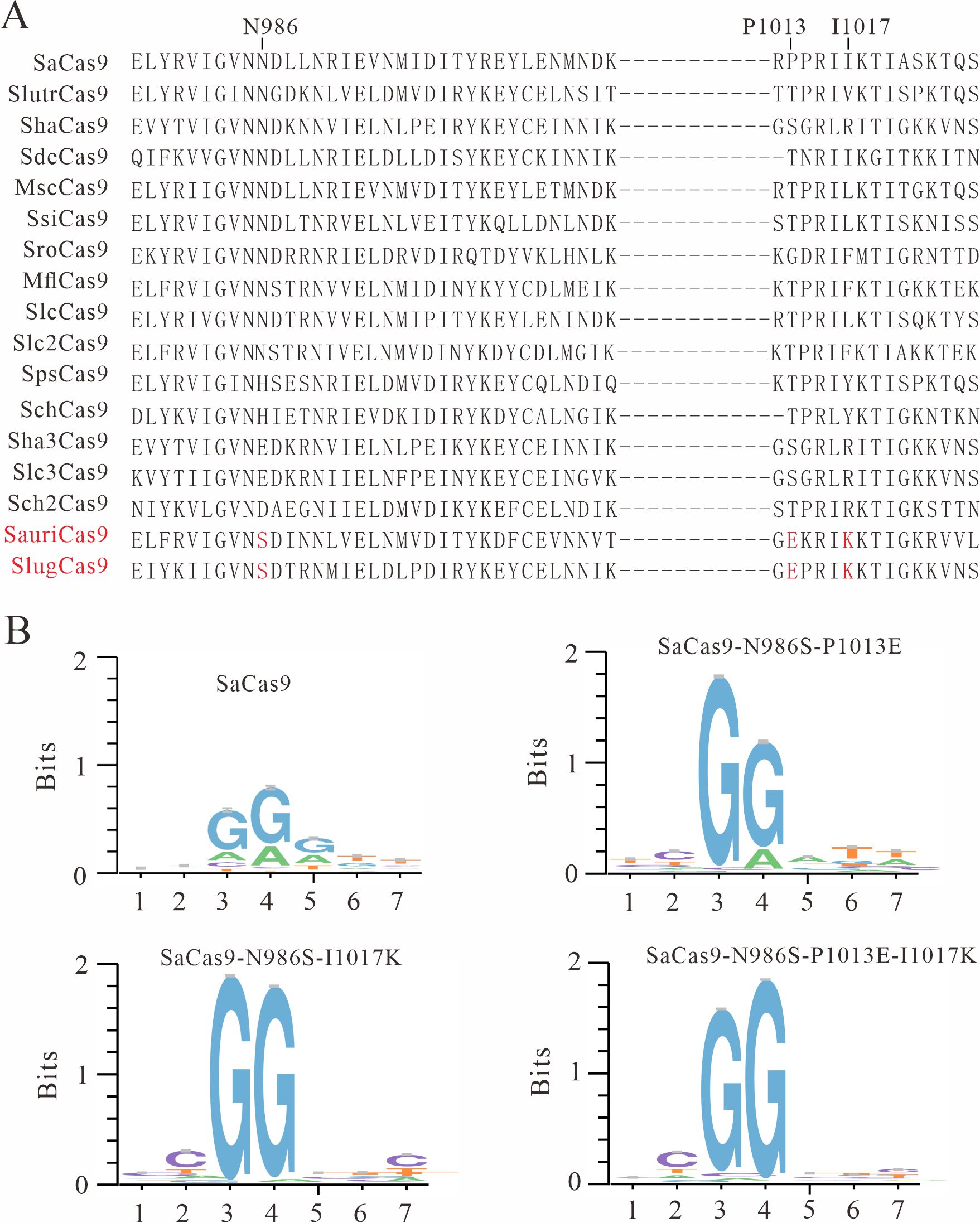
图1. (A)17个SaCas9同系物PI结构域的氨基酸序列。SauriCas9和SlugCas9特有的3个氨基酸用红色标记出来。(B)将SaCas9的2个或者3个氨基酸替换后识别NNGG PAM。
此外,该研究又在数据库中找到了5个含有保守氨基酸的Cas9,实验证实它们都识别NNGG PAM(图2)。

图2. (A)5个新的SaCas9同系物PI结构域的氨基酸序列。特有的氨基酸用红色标记出来。(B)5个SaCas9同系物的PAM序列。
Sha2Cas9和SpeCas9具有很高的编辑活性,但是存在脱靶现象。将Cas9和DNA之间形成非特异性氢键的氨基酸替换后,得到了精准版本的Cas9,极大的减少了脱靶(图3)。Sha2Cas9、SpeCas9和先前开发的SlugCas9都具有姘美SpCas9活性的优点。此外,还具有基因小、编辑范围大、不容易脱靶等优点,是基因编辑的理想工具。
王永明课题组致力于开发小型CRISPR-Cas9工具,建立了高效的筛选平台,先后开发出SauriCas9(NNGG PAM,PLoS Biol 2020)、SlugCas9(NNGG PAM,Nucleic Acids Res 2021)、SchCas9(NNGR PAM,Adv Sci 2022)、NarCas9(NNNNC PAM,eLife 2022)等,不断扩大编辑范围,提高编辑性能。

图3.(A)GFP报告基因分析脱靶。(B)GUIDE-seq全基因组分析脱靶。
复旦大学生命科学学院硕士生王帅和生陶晨是论文的共同第一作者,复旦大学王永明教授,东南大学柴人杰教授和苏州大学胡士军教授是论文的共同通讯作者。
原文链接:https://doi.org/10.1371/journal.pbio.3001897


