大量近期研究表明, 染色质在细胞核内的三维空间结构, 例如A/B区室(compartment), 拓扑关联结构域(TAD)和染色质环(chromatin loop), 可以在一定程度上调控细胞特异性的基因表达。其中染色质环结构富集于不同基因表达调控元件之间,对于深入了解转录调控机制具有重要助益。 目前,染色质构象捕捉技术(Hi-C)已被广泛应用于描绘染色质在细胞核内的组织分布以及鉴定染色质的三维特征结构。而近年来快速发展的单细胞染色质构象捕捉(单细胞Hi-C)技术更使得人们可以在单细胞水平研究染色质在细胞核内的结构和分布。然而现有的识别染色质环结构的算法大都是针对由大量细胞产出的且进行深度测序的Hi-C数据开发的,并不适用于极度稀疏的单细胞Hi-C数据。因此,亟需开发专门适用于单细胞Hi-C数据的识别染色质环的数据分析方法。
2021年8月26日,于淼青年研究员团队与美国克里夫兰诊所胡明博士团队和加州大学圣地亚哥分校任兵教授团队等合作在Nature Methods杂志在线发表了题为“SnapHiC: a computational pipeline to identify chromatin loops from single cell Hi-C data”的文章,报道了他们开发的名为SnapHiC的新算法,可从少量单细胞Hi-C数据中准确地识别高分辨率(文中为10Kb分辨率)的染色质环结构。SnapHiC软件目前已公开发布在Github网站(https://github.com/HuMingLab/snapHiC)。
SnapHiC算法首先利用重启随机游走算法(random walk with restart, RWR)对每个单细胞的染色质相互接触图谱进行补全, 由此来推测染色体上任意两个位点之间的接触概率。之后SnapHiC通过配对t-检验鉴定出相互接触概率显著高于预期的位点对,作为候选的染色质环结构。为了尽可能地降低假阳性的比例,SnapHiC要求由候选的染色质环所联结的两个位点无论是在全局或局部背景模型下,其相互作用概率均在统计学意义上显著高于预期值。最后,SnapHiC对所有候选染色质环进行聚类,并挑选出每一个聚类簇中相互作用最强的位点对作为最终识别的染色质环。
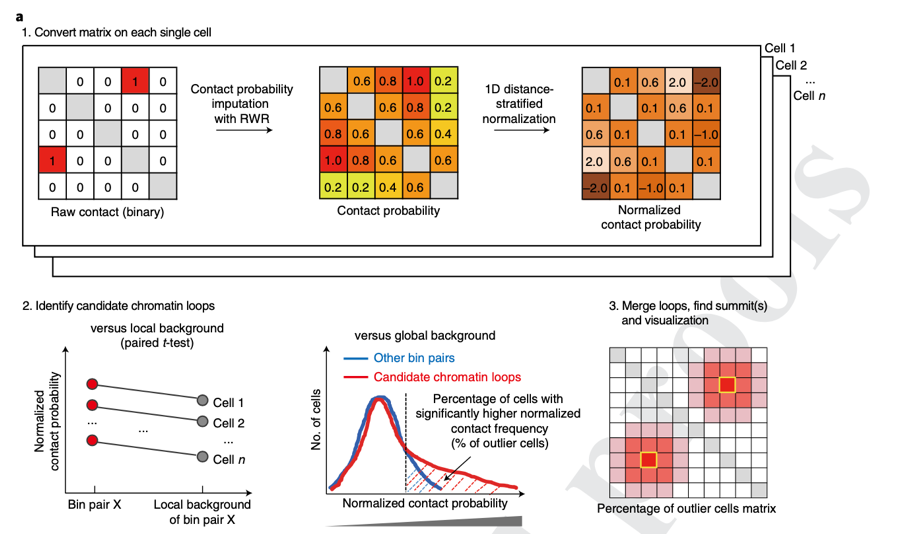
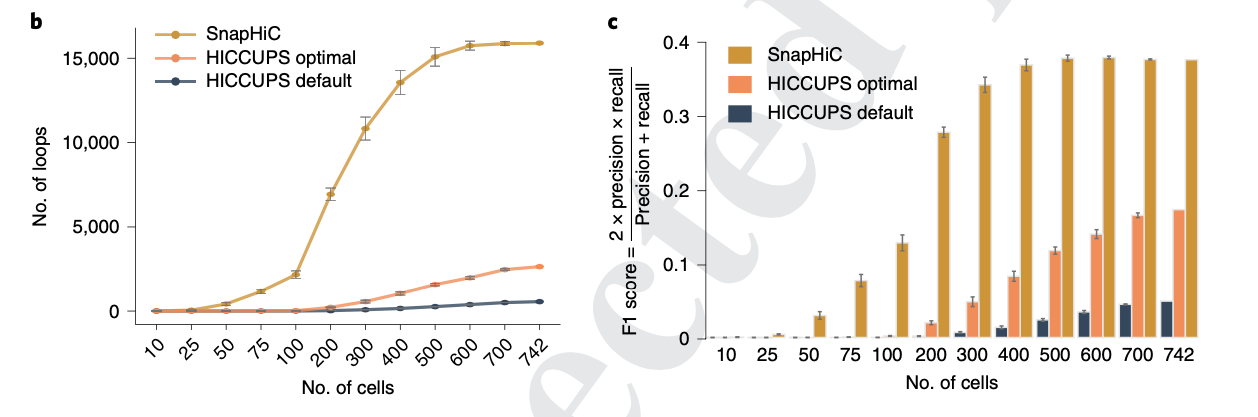
与传统的将多个单细胞Hi-C数据叠加模拟大量细胞的Hi-C图谱之后再识别染色质环的思路不同,SnapHiC将每个细胞视作独立的数据集,通过对单细胞核内染色质相互接触频率的方差进行估计,提高了统计学检验的效能,从而能够利用更少的单细胞Hi-C数据更准确地识别出更多的染色质环。这种统计学效能的提高,在细胞数较少时优势尤为明显。
作者首先将SnapHiC应用于已发表的小鼠胚胎干细胞的单细胞Hi-C数据集,并与目前大量细胞Hi-C数据分析中常用的多种染色质环识别算法(HiCCUPS、FastHiC、FitHiC2和HiC-ACT)进行比较,结果显示相较于传统算法,SnapHiC可在从更少的细胞中识别到更多更准确的染色质环。
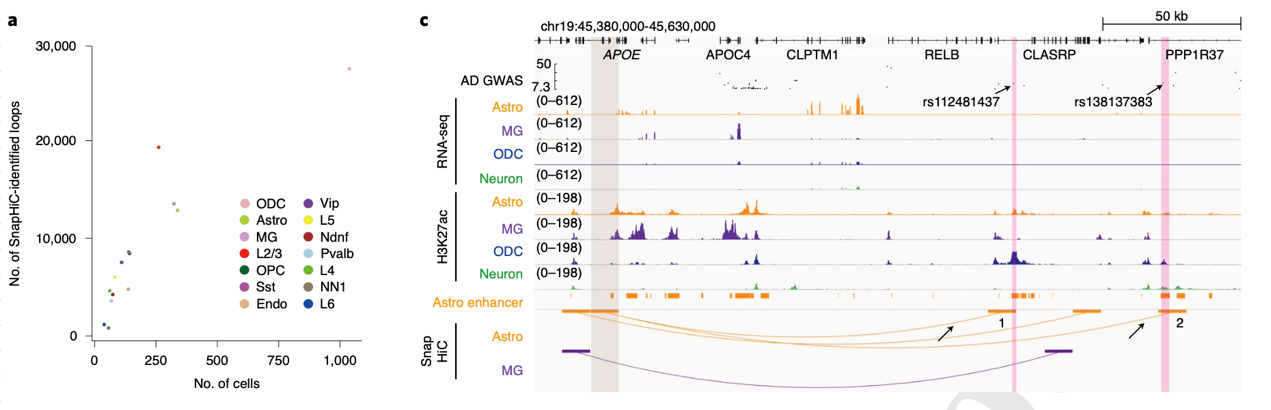
作者随后将SnapHiC应用于2019年发表的人脑前额叶皮层组织的单个细胞内的DNA甲基化和染色质三维结构 (sn-m3C-seq) 数据集 (PMID: 31501549)。作者从14种不同的细胞类型中, 更近一步地鉴定出每种细胞中的染色质环结构。分析结果发现很多染色质环具有细胞类型特异性,并且由这些细胞类型特异性的染色质环所联结的两端DNA通常与细胞特异性的基因表达与转录调控相关。作者最后利用这些染色质环将335个与神经精神障碍疾病相关的SNP位点与其可能调控的靶基因联系起来。例如在星形胶质细胞(astrocytes)中,2个位于增强子内的与阿尔兹海默症(Alzheimer’s disease)关联的序列变异(rs112481437, rs138137383) 与APOE基因的启动子区域形成染色质环结构,揭示了这两个SNP位点在星形胶质细胞中可能参与APOE基因的转录调控。 结果还显示该染色质环结构在星形胶质细胞中的特异性:因为该结构并没有在其它细胞类型中(如神经细胞neurons、小胶质细胞microglia和寡突胶质细胞oligodendrocytes)出现。
算法SnapHiC填补了利用单细胞Hi-C数据进行可靠染色质环鉴定方法领域的空白,极大的提高了从少量单细胞Hi-C数据中识别染色质环的检测效能,有助于未来更好地研究复杂组织中的不同细胞类型中特异性染色质高级空间结构。
于淼青年研究员和现任NovaSignal公司软件工程师的Armen Abnousi博士为本文的共同第一作者。美国克里夫兰诊所胡明博士和加州大学圣地亚哥分校任兵教授为该论文的共同通讯作者,复旦大学生命科学学院为第一单位。

