9月4日,我院遗传工程国家重点实验室麻锦彪教授课题组在核酸外切酶SDN1如何参与小非编码RNA周转代谢的研究中取得重要进展。该研究在分子水平阐明了SDN1识别剪切多种RNA底物的工作机制,并提出了SDN1在生物体内修剪小非编码RNA的分子模型,对于其他参与小非编码RNA降解代谢的核酸外切酶的可能作用方式提供了参考。相关研究成果以《拟南芥SDN1对于小RNA 3’端修剪机制的结构生化研究》(Structural and biochemical insights into small RNA 3’end trimming by Arabidopsis SDN1)为题在线发表于《自然·通讯》(Nature Communications)。
小非编码RNA是一类重要的调控RNA,一般参与到基因的转录后沉默中,这一过程也称为RNA干扰。微小RNA (miRNA)是其中重要的一类,其自身含量也需要受到精确的调控以维持稳态。拟南芥中的SDN1蛋白是一种特异性作用于小非编码RNA的核酸外切酶,通过剪切成熟miRNA的3’端核苷酸使其不稳定,进而进入周转途径。miRNA的降解周转对生物体进行正常代谢是必须的,拟南芥中SDN家族基因表达下调后会造成体内的miRNA水平上升,而植株会产生叶片变小、边缘锯齿化和早花等多种发育缺陷。
麻锦彪教授课题组通过X射线晶体学衍射和多种生物化学等手段解析了拟南芥SDN1的氮端结构域以及催化结构域与RNA底物的复合物的晶体结构,从分子水平阐释了SDN1活性催化口袋对于RNA底物的识别。并通过对SDN1的碳端RRM (RNA Recognition Motif)结构域的晶体结构的解析、核磁滴定以及一系列生化实验的证据,提出了RRM结构域在SDN1识别剪切单链或双链RNA底物时,与催化结构域协同作用的工作机理,即:SDN1通过RRM结构域结合单链RNA的5’端区域或者双链RNA中互补链的5’端单链区,而催化结构域识别并剪切RNA的3’末端。同时该研究表明SDN1与拟南芥AGO蛋白的PAZ结构域之间存在直接的相互作用,而这一相互作用可能促使miRNA 3’端脱离PAZ结构域的保护,进而被SDN1识别剪切,从而提供了在体内生理状态下SDN1如何作用于结合在AGO上的miRNA的可能机制。这一研究结果也为理解广泛参与不同小非编码RNA的生成和代谢过程的多种核酸外切酶的分子机制提供的一种可能的共同模型(示意图),也为用于实验室研究和作为核酸药物的更加稳定的小非编码RNA的设计和优化提供了基础。
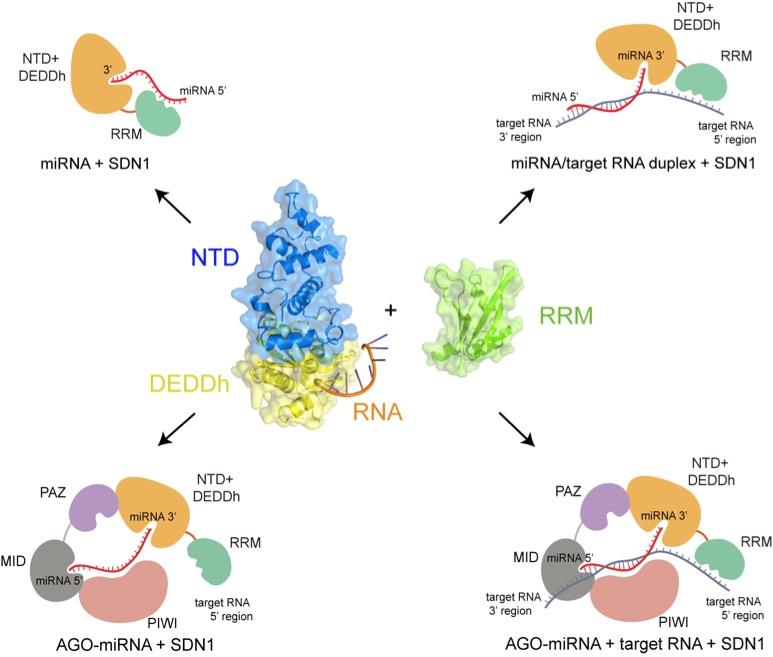
示意图:SDN1识别剪切不同类型植物小非编码RNA底物的可能机制模型
复旦大学生命科学学院和遗传工程国家重点实验室为该工作的第一完成单位,复旦大学生命科学学院生物化学系博士研究生陈嘉怡为本文第一作者。复旦大学生命科学学院和遗传工程国家重点实验室麻锦彪教授主持该项研究工作。加州大学河滨分校陈雪梅教授(Xuemei Chen)课题组参与了SDN1对于不同小RNA底物以及与结合在AGO上miRNA的体外生化分子机制研究,中科院上海生科院生化细胞所黄旲研究员参与了SDN1蛋白纯化、与RNA复合物的结晶以及晶体结构解析工作。该研究工作得到国家自然科学基金、中组部千人计划、遗传工程国家重点实验室的经费支持。

