6月12日,我院戚继课题组在《Methods in Ecology and Evolution》杂志在线发表题为“PhyloMCL: accurate clustering of hierarchical orthogroups guided by phylogenetic relationship and inference of polyploidy events”的研究论文,该算法为基因组尺度上研究真核生物间的同源基因提供了准确高效的新工具。
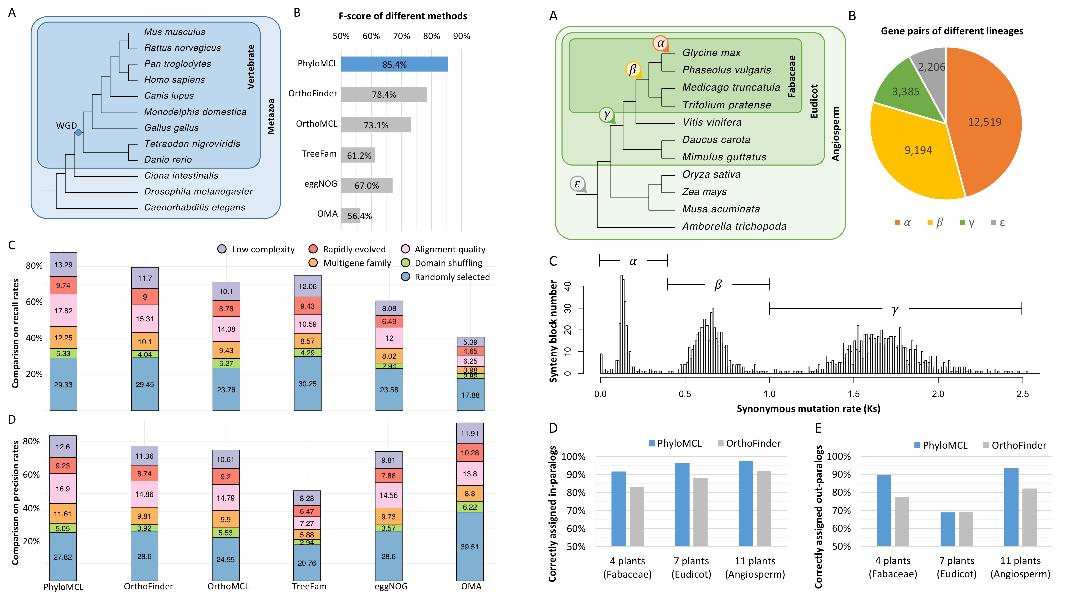
图. PhyloMCL与同类方法在动物和植物基因组中的分析结果比较
准确判断物种间同源基因的相互关系对于推断基因的分子生物学功能、大尺度重建基因家族的系统发育关系、重建真核生物各类群的进化历史至关重要。同源基因识别与聚类往往是分子遗传学和基因组学研究的必要步骤。动植物基因组中存在大量的重复基因,极大增加了基因组的复杂性。一方面重复基因通过累积随机突变发生新功能化和亚功能化,为新基因和新功能的产生提供了源泉,有利于促进增加物种的环境适应性;另一方面,重复的存在也对如何准确识别同源基因的生物信息学方法开发提出了挑战,难点包括同源基因突变速率异质性、基因融合和基因分裂等复杂进化事件。特别是脊椎动物祖先、被子植物祖先和内部类群发生的染色体多倍化事件进一步增加了正确判断同源基因亲缘关系的困难程度。
该研究提出了一种基于马尔可夫基因聚类的生物信息学新方法PhyloMCL(https://phylomcl.sourceforge.io/),纳入了生物类群的系统发育关系作为物种框架参考,并充分考虑了不同分类层次的染色体多倍化对重复基因保留程度的影响,可以在多物种的全基因组规模上准确识别基因的直系和旁系同源关系,并以层次同源聚类的方式展现具体结果。为验证方法的有效性,该文章分别采用动物和植物的多组基因组进行准确性评估:(1)将该方法应用到12个后生动物的基因组聚类分析时,召回率和准确率分别达到87.8%和83.2%,大幅超过已发表的同类研究方法;(2)该方法在分析25个种子植物基因组时,可以准确识别初被子植物进化过程中的多次多倍体事件。该方法进一步将个体植物基因组中数以万计的旁系同源基因划分为不同的演化起源,成功识别出各分类层次发生的内/外旁系同源基因,准确性超过90%。PhyloMCL得益于Markov聚类策略和物种树指导,可以在可承受的时间内准确地对数百万个同源基因进行分类,从而满足了测序基因组快速增长带来的植物学研究挑战。
综上所述,PhyloMCL方法采用马尔可夫聚类策略并充分纳入物种亲缘框架和多倍化因素的考量,可以在同时对上百个基因组中数以百万级别的同源基因进行准确高效的分类,解决了基因组高速增长为分子遗传学和基因组学带来的挑战。
我院16级博士生周盛予为本研究的第一作者,遗传工程国家重点实验室的戚继研究员和江西农业大学的国春策教授为文章的共同通讯作者。该研究获得国家自然科学基金和复旦大学遗传工程国家重点实验室的共同资助。
全文链接:https://besjournals.onlinelibrary.wiley.com/doi/full/10.1111/2041-210X.13401

