近日,我院遗传工程国家重点实验室洪尚宇课题组与中科院上海生化细胞所廖侃研究员和陈正军研究员课题组合作在《尖端科学》(Advanced Science)上在线发表题为《Cavin1缺失影响小鼠肝血窦内皮细胞的窗孔形成并导致肝糖原代谢异常与新生死亡》(Cavin1 deficiency causes disorder of hepatic glycogen metabolism and neonatal death by impacting fenestrations in liver sinusoidal endothelial cells)的研究论文1。该研究发现Cavin1基因(又称PTRF基因)对于小鼠肝血窦内皮细胞上的窗孔结构至关重要,敲除Cavin1基因会大幅减少窗孔数量、抑制小鼠肝实质细胞与血液的物质交换,从而破坏糖原与血糖的平衡关系并导致新生小鼠低血糖和死亡。
在人体中,CAVIN1基因的纯合突变会导致一种非常罕见的常染色体隐性遗传疾病:先天性脂肪营养不良4型(CGL4)2,3。CGL4患者主要表现为全身性的脂肪细胞缺失和肌肉萎缩,后期常伴有胰岛素抵抗、糖尿病和高甘油三脂血症2,3。目前,有报道的CGL4患者仅有约30例,因此研究极少。Cavin1的全身性敲除小鼠表现出与CGL4病人相似的病理特征4。因此研究Cavin1敲除小鼠可以揭示Cavin1的功能和分子机制,为CGL4患者提供可能的治疗或关怀。
在研究过程中,洪尚宇课题组与合作团队发现Cavin1敲除小鼠的肝血窦内皮细胞上的窗孔结构的密度比野生型小鼠显著减少。窗孔的数量和直径变化都会影响葡萄糖,脂质,蛋白质和其他分子通过内皮细胞形成的血管屏障到达肝实质细胞5-7。因此,这些窗孔结构决定了肝血窦内皮细胞的通透性,对肝脏与血液之间的物质交换至关重要。而新生的Cavin1敲除小鼠出现明显的肝脏糖原储存和分解异常,并引起血糖调控紊乱。大部分敲除小鼠在出生后数小时内即出现严重的低血糖症并死亡。如果在小鼠出生后立刻注射葡萄糖则能够帮助Cavin1敲除小鼠度过这段血糖缺乏时期,直到它们可自行获得母乳并存活。
近年来,越来越多的科学家关注到肝血窦内皮细胞上的窗孔结构在衰老、营养状况(过度饮食、饥饿)或疾病(肝硬化、癌症)等生理和病理过程中的重要作用8,9,但调节窗孔结构的分子机制仍然未知。洪尚宇课题组与合作团队通过研究发现Cavin1敲除能够抑制RhoA-ROCK2-LIMK-Cofilin信号通路并抑制细胞骨架的动力学,并最终导致肝血窦内皮细胞上窗孔的减少;而注射微丝解聚剂红海绵毒素A则能够增加Cavin1敲除小鼠肝血窦内皮细胞上的窗孔数目,并改善Cavin1敲除小鼠的新生小鼠存活率。因此,Cavin1与细胞骨架动力学是肝血窦内皮细胞上窗孔形成与维持的重要机制。
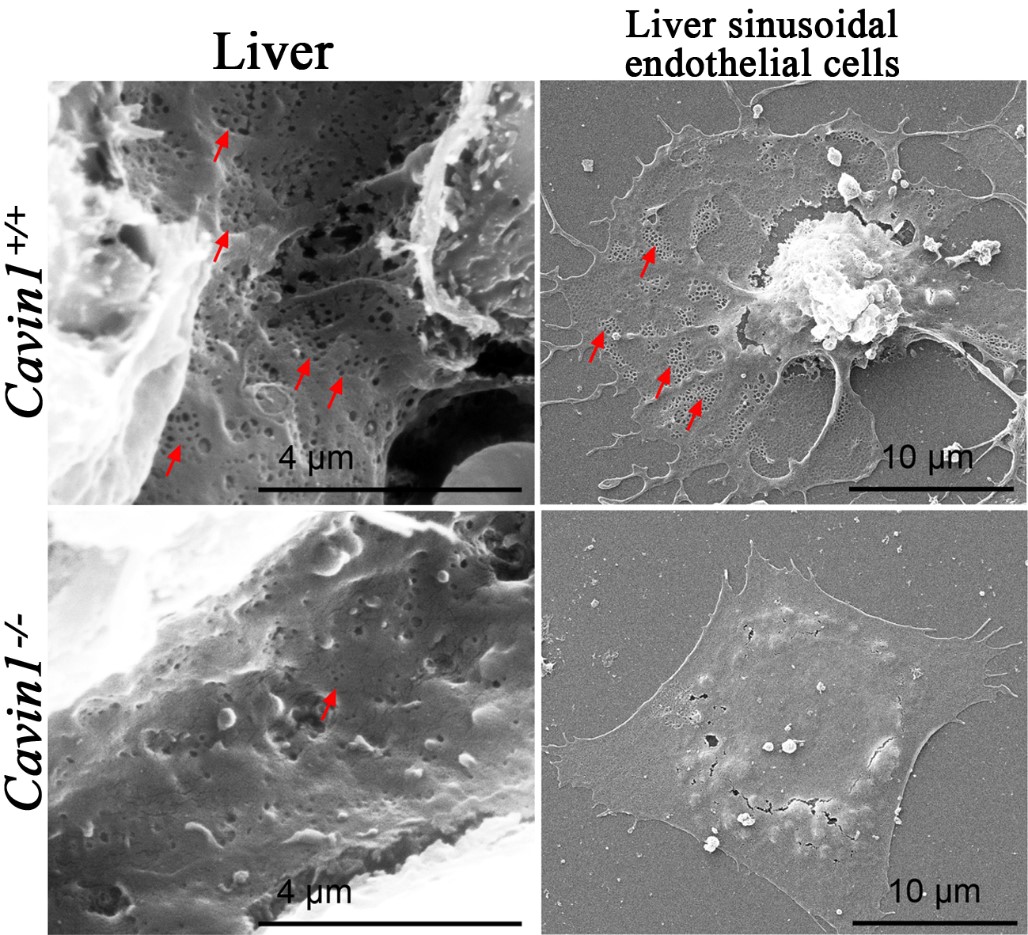
图示:Cavin1敲除导致肝血窦内皮细胞窗孔减少。左图为小鼠肝组织扫描电镜图;右图为体外培养的原代肝血窦内皮细胞的扫描电镜图。
红色箭头所指为窗孔结构。
论文还指出,Cavin1敲除小鼠的新生死亡仅在某些遗传背景下(如纯C57BL / 6J背景)发生。而在C57BL / 6J和 FVB(各50%)的杂交背景下,Cavin1敲除小鼠的存活率与野生型小鼠相比无显著差异1。这可能也是为什么迄今尚未有患有CGL4的新生儿死亡的原因之一,在人群中,新生儿死亡有可能会且只会发生在具有一定基因背景的少数CGL4患者中。无论如何,我们都应密切监测CGL4患者新生儿的血糖水平,防止严重低血糖导致的新生儿死亡。
魏庄、雷继刚、戴宇翔以及沈丰为论文的共同第一作者。复旦大学洪尚宇与中科院上海生化细胞所廖侃、陈正军为共同通讯作者。复旦大学为第一单位。该研究得到了科技部国家重点研发计划、国家自然科学基金委重大研究计划以及上海市科技重大专项、浦江人才计划的支持。
全文链接:https://onlinelibrary.wiley.com/doi/10.1002/advs.202000963
参考文献:
1. Z. Wei, J. Lei, F. Shen, Y. Dai, Y. Sun, Y. Liu, Y. Dai, Z. Jian, S. Wang, Z. Chen*, K. Liao* and S. Hong, Advanced Science, 2020, https://doi.org/10.1002/advs.202000963.
2. N. Patni and A. Garg, Nature reviews. Endocrinology, 2015, 11(9), 522-534.
3. G. Nilay, T. Kutlu, G. T. Tekant, A. G. Eroglu, N. C. Ustundag, B. Ozturk, H. Onay, and B. Tuysuz, European journal of medical genetics, 2019, 103819.
4. L. Liu, D. Brown, M. McKee, N. K. Lebrasseur, D. Yang, K. H. Albrecht, K. Ravid, and P. F. Pilch, Cell metabolism, 2008, 8(4), 310-317.
5. F. Braet and E. Wisse, Comparative hepatology, 2002, 1(1), 1.
6. F. Braet, Liver international: official journal of the International Association for the Study of the Liver, 2004, 24(6), 532-539.
7. F. Braet, M. Shleper, M. Paizi, S. Brodsky, N. Kopeiko, N. Resnick, and G. Spira, Comparative hepatology, 2004, 3(1), 7.
8. R. V. Stan, D. Tse, S. J. Deharvengt, N. C. Smits, Y. Xu, M. R. Luciano, C. L. McGarry, M. Buitendijk, K. V. Nemani, R. Elgueta, T. Kobayashi, S. L. Shipman, K. L. Moodie, C. P. Daghlian, P. A. Ernst, H. K. Lee, A. A. Suriawinata, A. R. Schned, D. S. Longnecker, S. N. Fiering, R. J. Noelle, B. Gimi, N. W. Shworak, and C. Carriere, Developmental cell, 2012, 23(6), 1203-1218.
9. A. Hammoutene and P. E. Rautou, Journal of hepatology, 2019, 70(6), 1278-1291.

