2025年10月1日,复旦大学生命科学学院田卫东教授课题组在 Human Reproduction期刊在线发表了题为 “Identifying candidate genes for spermatogenic failure and predicting ICSI outcomes using single-cell RNA sequencing and protein-protein interaction networks” 的研究论文。该研究创新性地联合单细胞转录组(scRNA-seq)与蛋白互作网络(PPI)构建机器学习框架,实现了精子发生障碍(spermatogenic failure)相关基因的系统识别及ICSI(卵胞质内单精子注射)临床结局的精准预测,为男性不育的遗传诊断和辅助生殖决策提供了全新思路。

研究背景
精子发生障碍是男性不育的主要原因之一。然而,现有诊断手段主要依赖精液分析,难以揭示其潜在的遗传机制。尽管OMIM数据库已收录百余个相关致病基因,但临床分子诊断率仍然较低。此外,虽然ICSI技术已成为治疗精子发生障碍的常用手段,但不同遗传背景下的治疗成功率差异显著。因此,系统识别精子发生障碍相关基因并预测ICSI生殖结局,对于提高分子诊断率和辅助个体化治疗具有重要的临床意义。
模型设计
研究团队构建了结合单细胞转录组与蛋白互作网络的机器学习框架。该框架包含三类模型:
模型一(基因预测模型):基于81种人类细胞类型的scRNA-seq表达特征与PPI网络嵌入信息,筛选与精子发生障碍相关的候选基因。
模型二(亚型分类模型):结合精子发育轨迹(B样条特征)与PPI特征,实现不同表型亚型(无精子症、畸形精子症、弱精子症)的区分。
模型三(ICSI结局预测模型):在候选基因层面上预测ICSI临床结局。
模型采用随机森林算法,并经五折交叉验证及多组独立数据集验证,表现出优异的预测性能(例如ICSI结局预测模型PRC-AUC=0.96,ROC-AUC=0.82)。
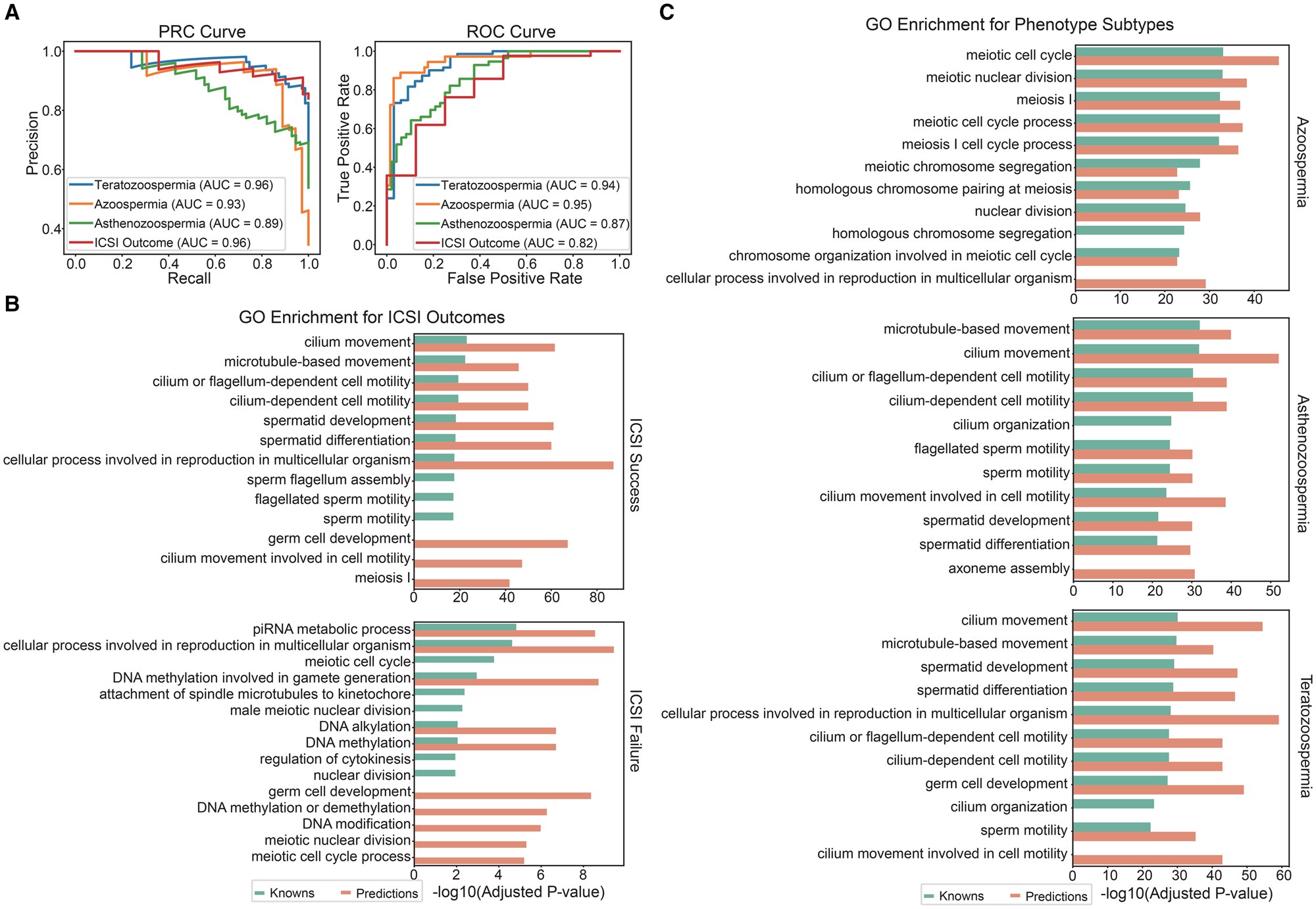
图:精子发生障碍亚型与ICSI结局的预测
核心发现
系统识别精子发生障碍候选基因:模型预测出320个潜在致病基因,其中61.6%已有文献患者或小鼠模型支持,验证了模型可靠性。
亚型特异性功能模块:通过加权基因共表达网络分析(WGCNA)与PPI聚类,发现不同亚型间的基因存在显著分化,揭示其分子异质性。
ICSI结局预测与临床验证:在34例精子发生障碍患者的全外显子组测序(WES)数据中,模型辅助分析将分子检出率由11.8%提升至29.4%。携带候选基因(如 TDRD12、HFM1、RAD51AP2、GARIN2)突变的个体,其表型亚型和ICSI结局与模型预测结果高度一致。
研究意义
该研究将单细胞转录组、蛋白互作网络与机器学习深度融合,实现了从基因优先排序到临床结局预测的全流程建模。研究不仅揭示了精子发生障碍的潜在致病机制,也为个体化辅助生殖治疗提供了计算工具。该模型框架已开源发布(https://github.com/TianLab-Bioinfo/SpermGenePred),为后续精准生殖医学研究提供了可复用的范例。
复旦大学生命科学学院计算生物学系博士生刘浏为本文第一作者, 田卫东教授为通讯作者。该研究得到了国家自然科学基金、上海市市级科技重大专项及国家重点研发计划的支持。
论文链接:https://doi.org/10.1093/humrep/deaf186

