2026年2月16日,复旦大学生命科学学院董爱武教授课题组、刘兵青年研究员课题组与甘建华研究员课题组合作,在《自然通讯》(Nature Communications)在线发表了题为“H3K4me1 directs H3K36me2 and H3K36me3 deposition in land plants”的研究论文。该研究首次揭示了H3K4me1修饰在陆生植物中的独特功能:H3K4me1可被特定阅读蛋白所识别,进而招募H3K36甲基转移酶,促进H3K36me2和H3K36me3修饰的建立。
组蛋白翻译后修饰(post-translational modifications, PTMs)在转录调控中发挥关键作用,并广泛参与真核生物生长发育、环境响应等多种生物学过程。PTMs由特定的“写入”酶和“擦除”酶动态调控,并可被不同的“阅读”蛋白所识别。其中,组蛋白H3第4位赖氨酸(H3K4)甲基化是研究最为深入的修饰之一,其不同修饰状态(单甲基化、二甲基化、三甲基化)可分别标记转录激活或抑制。哺乳动物中,H3K4单甲基化(H3K4me1)修饰已被证实是增强子的标记,然而其在植物中的功能尚不明确。
为解答上述科学问题,选取了单细胞水生物莱茵衣藻、早期登陆物小立碗藓以及开花植物拟南芥和水稻,进行表观基因组分析,发现陆生植物中H3K4me1在全基因组范围内的分布模式及其与基因转录的相关性均呈现出独特的特征,提示H3K4me1在陆生植物中可能具有不同于其他生物的功能(图a)。为深入揭示H3K4me1在陆生植物中的功能,筛选到特异性识别H3K4me1的阅读蛋白Ehd3(Early heading date 3)。晶体结构解析表明,Ehd3通过其串联PHD结构域(tandem PHD finger domain)特异识别H3K4me1修饰,该识别模式与已知的PHD结构域蛋白完全不同(图b)。功能回复实验显示,突变Ehd3中识别H3K4me1的关键氨基酸无法挽救ehd3-1突变体的晚花表型,说明Ehd3对H3K4me1的识别为其发挥生物学功能所必需。进一步通过IP-MS等技术,鉴定到Ehd3的结合蛋白H3K36甲基转移酶SDG724(SET domain group 724)(图c)。表型与组学分析发现,Ehd3或SDG724缺失均导致植物在生长发育、转录组及表观基因组层面表现出相似的变化。整合Ehd3、SDG724和组蛋白修饰的ChIP-seq结果,发现Ehd3、SDG724与H3K4me1修饰在全基因组范围共定位;在ehd3-1,sdg724-1及ehd3 sdg724突变体中,H3K36me2/me3的全基因组分布发生一致性的全局改变(图d)。体外酶活实验进一步证明,SDG724的H3K36甲基转移酶活性依赖于Ehd3和H3K4me1的存在(图e)。最后,跨物种组学分析显示,在陆生植物中H3K4me1与H3K36me2/me3在全基因组水平上具有显著的共定位特征。综上所述,本研究系统阐明了H3K4me1在陆生植物中介导H3K36me2/me3建立的分子机制,揭示了H3K4me1在植物中具有与动物中完全不同的功能。
绍兴大学生命与环境科学学院讲师吴佳炳、复旦大学董爱武教授团队博士研究生王嘉琛、四川农业大学水稻研究所副教授杜康兮以及泰州学院药学院讲师李颖平为本文共同第一作者。复旦大学生命科学学院董爱武教授、刘兵青年研究员及甘建华研究员为共同通讯作者。复旦大学生物医学研究院蓝斐实验室对本工作提供了重要支持。中国科学院上海高等研究院上海光源(SSRF)科学中心、国家蛋白质科学研究(上海)设施规模化蛋白质制备系统为本研究提供了技术支持。本研究得到了国家重点研发计划、国家自然科学基金和西南作物基因资源发掘与利用国家重点实验室开放课题项目的资助。
全文连接:https://doi.org/10.1038/s41467-026-69632-5
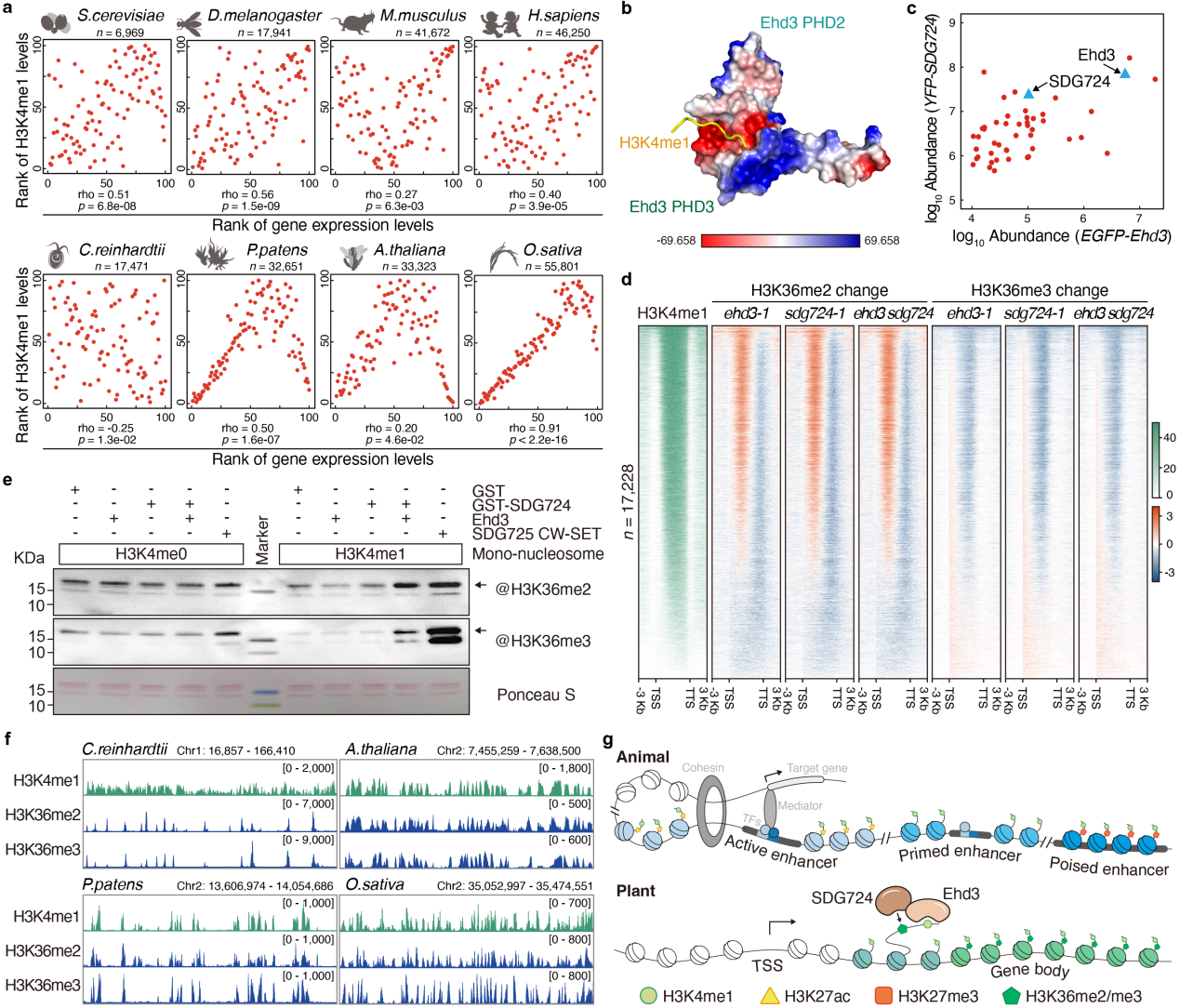
a.陆生植物中H3K4me1与基因表达呈现出独特的相关性; b. Ehd3串联PHD结构域识别H3K4me1肽段的晶体结构;c. Ehd3与H3K36甲基转移酶SDG724存在相互作用; d.Ehd3或SDG724缺失导致H3K36me2/me3的分布模式发生全局变化; e.酶活实验证实SDG724的甲基转移酶活性依赖于Ehd3和H3K4me1; f. 陆生植物中H3K4me1与H3K36me2/me3共定位; g. H3K4me1在动、植物中具有不同的功能。

