近期,生物传感器领域权威学术期刊《生物传感器和生物电子学》(Biosensors & Bioelectronics) 在线发表了复旦大学生命科学学院、遗传工程国家重点实验室黄强教授课题组关于核酸适配体分子设计的研究论文:De novo design of DNA aptamers that target okadaic acid (OA) by docking-then-assembling of single nucleotides。该论文开发了一种核酸适配体从头设计新方法—单核苷酸对接后组装(Docking-then-assembling of single nucleotides)方法,并用该方法设计了靶向海洋生物毒素冈田酸(Okadaic acid, OA)的高亲和性和高特异性的DNA适配体,在核酸适配体设计方面取得了重要结果。
生物传感器具有灵敏度高、特异性强、操作简单等特点,广泛用于生物分子检测,在食品安全等方面有重要意义。作为生物传感器的识别元件,核酸适配体在合成速度、成本等方面具有明显优势。然而,目前用于核酸适配体筛选的SELEX (Systematic Evolution of Ligands by Exponential Enrichment,指数富集配体系统进化) 技术随机性大,需要合成和测序大量候选适配体,耗时耗力。因此,开发针对特定靶标分子的适配体精准设计方法将有助于大大降低获取高亲和性核酸适配体的成本。
在研究中,黄强教授课题组运用计算结构生物学技术并借鉴药物分子片段设计的理念,创新性地提出了“单核苷酸对接后组装”的核酸适配体从头设计方法,成功地设计了冈田酸的高亲和性和高特异性核酸适配体。单核苷酸对接后组装方法主要包括三个步骤:首先,采用单核苷酸分子对接识别靶分子上的高亲和性结合位点;接着,把结合位点上对接所得的核苷酸组装为短片段,作为核酸适配体的结合单元;最后,以热稳定性好的二级结构(如迷你DNA发夹结构)为适配体的稳定单元,与结合单元共同组装成全长适配体。经微量热泳动实验验证,研究所设计的5条亲和性适配体的平衡解离常数 (Kd) 值在100-600 nM之间,且亲和性最高的适配体对冈田酸结合具有特异性,与以往用SELEX方法大规模实验筛选所得的适配体相当,但所需成本大大降低,充分体现了新方法的优势。因此,本项研究不仅为冈田酸检测用适配体传感器的开发提供了新的亲和性识别分子;而且,单核苷酸对接后组装方法是一种通用设计技术,可用于快速地精准设计特定靶标分子的高亲和性核酸适配体。
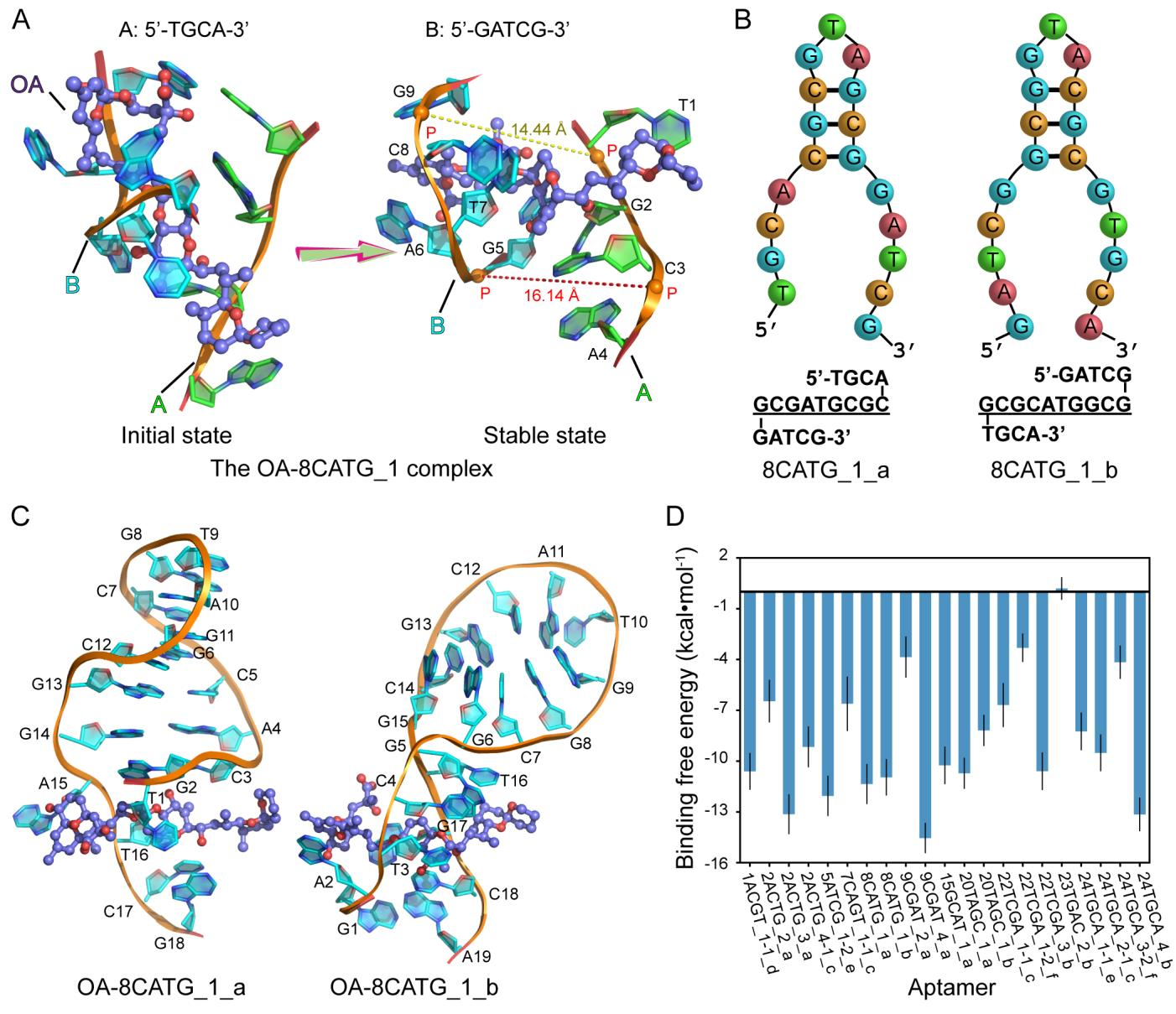
图1. 用单核苷酸对接后组装方法设计的冈田酸核酸适配体
生命科学学院博士研究生宋梦华同学为论文的第一作者,黄强教授为通讯作者,李园园同学、高瑞华博士和刘建平副教授参与了研究工作。论文研究获得了国家科技重大专项等项目的支持。课题组依托重点实验室的高性能集群计算机,及使用课题组建立的生物分子建模与设计平台完成了相关研究工作。
论文链接为:https://www.sciencedirect.com/science/article/pii/S0956566322006029

